|
 |
| 本日、大阪大学では、離れた場所に分散している複数の異機種の高速コンピュータをインターネットで結び、複雑な分子計算を高速処理するグリッド計算技術を開発したことを発表いたしました。 |
 |
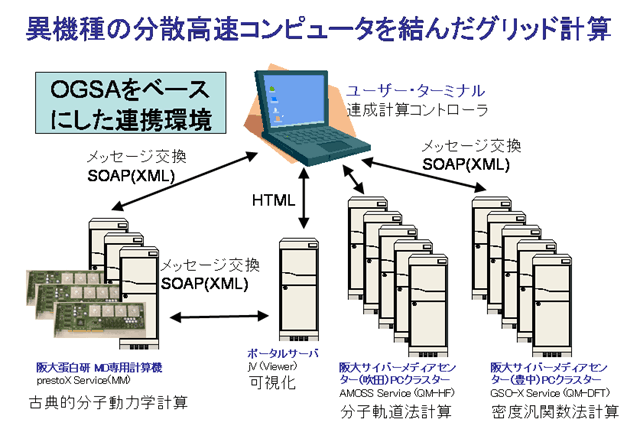
| 大阪大学サイバーメディアセンター(豊中キャンパス)の156CPUのPCクラスター、同センター(吹田キャンパス)の40CPUのPCクラスター、さらに同大学蛋白質研究所の分子動力学(MD)専用ボードMD-grape2を搭載したMD専用計算機の3種類の高速コンピュータを結び、それぞれNEC基礎・環境研究所量子化学計算グループ(高田俊和主席研究員)開発のプログラム
AMOSS (Ab initio Molecular Orbital System for Supercomputers)によるハートレー・フォック方程式を解く分子軌道法計算、大阪大学理学研究科化学専攻・山口兆教授らが開発したプログラムGSO-X(Generalized
Spin Orbital calculation-X)によるDFT(密度汎関数法)計算、さらに同大学蛋白質研究所と独立行政法人・産業技術総合研究所生物情報解析研究センター(中村春木教授・チームリーダー)開発のプログラムprestoX
(Protein Engineering Simulator eXtended)による古典論による分子動力学計算を別々に行いながら連成させ、長大な分子計算をグリッド計算システム上で実行する仕組み「バイオフーガ
(BioPfuga: Biosimulation Platform United on Grid Architecture)」を開発しました。 |
 |
| 各プログラムは、それぞれ別々のグループが数年〜10年以上の年月をかけて開発してきたものですが、個々のプログラムのコンポーネント化を進め、OGSA(Open
Grid Service Architecture) に基づく国際標準規格に沿ったGT3 (Globus Toolkit
version 3.2) によりWeb service化しました。この標準的なグリッド・アーキテクチャーを用いることにより、少ない開発工程にもかかわらず、異機種を結んだ高速グリッド計算が可能となりました。このように3種類の巨大な計算プログラムを連成させ、離れた場所に分散している計算機をつないで同時に稼働させたのは、世界でも初めての例です。 |
 |
| これらシミュレーション計算でやりとりする中間データは巨大なものとなるため、従来はバイナリー形式で中間データが記述される場合が多く、標準化の妨げとなっていました。今回は、bmsML
(Biomolecular Simulation Markup Language)というXML (eXtensible
Markup Language) によるデータ記述法も合わせて開発し、バイナリー形式のわずか1.3倍程度の大きさで異なるプログラムで中間データの授受が可能な仕組みとしました。 |
 |
| 蛋白質等の生体高分子における化学反応や分子認識反応の理解には、複雑な蛋白質全体のダイナミックな構造と同時に、局所機能部位における電子状態の詳細な解析が必要です。従来は、機能部位のみをモデル化した低分子に対する研究が主流でしたが、電子状態と蛋白質分子全体の状態を同時に解析する連成シミュレーション技術の必要性が高まっておりました。今回開発した連成計算システムの利用により、酵素反応やチャネル蛋白質分子機能の詳細な解析と分子設計が行われます。さらに、フィジオーム等マルチスケールのシミュレーション計算が注目されてる生体・細胞シミュレーション技術への応用も期待されています。なお、この計算プログラムシステムは、本年1月に認証・創設されたNPO法人「バイオグリッドセンター関西」を通じ、大学や企業研究者への利用に供される予定です。 |
 |
| 本研究開発は、文部科学省の研究開発委託事業であるITプログラム「スーパーコンピュータネットワークの構築」(研究代表者・下條真司大阪大学教授)により、大阪大学サイバーメディアセンター、大阪大学蛋白質研究所、大阪大学大学院理学研究科、NEC基礎・環境研究所、(株)日立製作所によって実施されました。 |
 |
 |